CRISPR para niños
Datos para niños CRISPR |
|
|---|---|
 |
|
| CRISPR + fragmentos de ADN de E.Coli. | |
| Identificadores | |
| Organismo | |
| Símbolo | ? |
| Otros datos | |
CRISPR es el nombre de unas secuencias especiales de ADN que se encuentran en organismos muy pequeños llamados procariotas (como las bacterias y las arqueas). Estas secuencias son como un "archivo de memoria" de ataques pasados de virus que intentaron infectar a estos organismos.
Las bacterias y arqueas guardan pequeños trozos del ADN de esos virus invasores en su propio código genético. Luego, usan estos trozos para reconocer y destruir a virus similares en el futuro. Es como un sistema inmunitario muy inteligente y específico que les permite defenderse.
Además de ser una defensa natural, estas secuencias CRISPR han dado origen a una tecnología muy avanzada llamada CRISPR/Cas. Esta tecnología permite a los científicos modificar los genes de cualquier organismo. Utiliza unas "tijeras moleculares" llamadas enzimas Cas (como la Cas9) que pueden cortar el ADN en lugares específicos para cambiar la información genética.
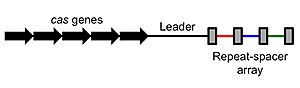
En resumen, los sistemas CRISPR son partes del ADN que tienen secuencias cortas que se repiten. Después de cada repetición, hay pequeños segmentos de "ADN espaciador". Estos espaciadores son recuerdos de virus que atacaron antes. Se encuentran en casi la mitad de las bacterias y en la mayoría de las arqueas. A menudo, están junto a los genes "cas", que producen las proteínas Cas, las "tijeras" que cortan el ADN.
El sistema CRISPR/Cas es una defensa para los organismos procariotas. Les ayuda a resistir a elementos externos como los plásmidos (pequeños círculos de ADN) y los bacteriófagos (virus que atacan bacterias). Funciona como una inmunidad adquirida, lo que significa que aprenden de ataques anteriores. Los espaciadores de CRISPR reconocen secuencias específicas y guían a las proteínas Cas para cortar y desactivar el ADN de los virus invasores.
Desde 2013, la tecnología CRISPR/Cas se usa para la ingeniería genética. Esto significa que se pueden añadir, quitar o cambiar partes específicas de los genes. También se puede usar para controlar la actividad de los genes. Al introducir la proteína Cas9 y unas moléculas guía especiales (llamadas ARN guía) en una célula, se puede cortar el genoma de esa célula en los puntos deseados. Esto permite a los científicos estudiar qué pasa cuando un gen no funciona o cuando se introduce un cambio. También se están desarrollando formas de usar CRISPR/Cas9 para solo modificar cómo funcionan los genes, sin cambiar su información genética. Los investigadores creen que CRISPR podría usarse para alterar los genomas de poblaciones enteras de organismos.
Contenido
Descubrimiento y desarrollo de CRISPR
Las bacterias tienen la capacidad de incorporar ADN de su entorno, incluso si está dañado.
Primeros hallazgos y el nombre CRISPR
Las secuencias repetidas que hoy conocemos como CRISPR fueron vistas por primera vez por científicos japoneses en 1987, liderados por Yoshizumi Ishino. Luego, a principios de los años 90, el científico español Francisco J. M. Mojica de la Universidad de Alicante las encontró de forma independiente en una arquea llamada Haloferax mediterranei. Sus resultados se publicaron en 1993.
Al principio, estas secuencias se usaban para identificar y diferenciar distintos tipos de bacterias. En el año 2000, Francisco J. M. Mojica y su equipo encontraron muchas de estas secuencias repetidas en bacterias, arqueas y mitocondrias. Propusieron llamarlas "Repeticiones Cortas Regularmente Espaciadas". Pocos años después, Mojica propuso y creó el nombre CRISPR (del inglés: Clustered Regularly Interspaced Short Palindromic Repeats), que significa "repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas". Este nombre fue adoptado universalmente en 2002. En esa misma publicación, se describieron por primera vez los genes "cas" (asociados a CRISPR), que producen proteínas relacionadas con estas secuencias.
En 2005, tres grupos de investigación diferentes descubrieron que algunos de los espaciadores de CRISPR provenían de ADN de fagos y de otros elementos genéticos. Fue el grupo de Francisco J. Mojica quien primero se dio cuenta de que las secuencias CRISPR y sus espaciadores podían ser parte de un sistema de defensa de estos microorganismos. Estas observaciones clave indicaron que el sistema CRISPR/Cas podría funcionar como una inmunidad adquirida en las bacterias.
En 2007, científicos de la industria alimenticia y de la Université Laval en Canadá demostraron que podían cambiar la resistencia de la bacteria Streptococcus thermophilus a los ataques de fagos usando ADN espaciador.
El papel de Cas9
Jennifer Doudna y Emmanuelle Charpentier, quienes ganaron el Premio Nobel de Química en 2020, junto con Francisco Juan Martínez Mojica, investigaron las proteínas asociadas a CRISPR para entender cómo las bacterias usan los espaciadores en su sistema de defensa. Estudiaron un sistema CRISPR más sencillo que usa una proteína llamada Cas9. Descubrieron que las bacterias responden a un virus invasor copiando los espaciadores y el ADN repetido en una molécula larga de ARN. Luego, la célula usa un ARN especial llamado tracrRNA y la proteína Cas9 para cortar esa molécula en pedazos más pequeños llamados ARNcr.
Cas9 es una nucleasa, una enzima que corta el ADN. Tiene dos puntos de corte activos, uno para cada hebra de la doble hélice del ADN. El equipo de investigación demostró que podían desactivar uno o ambos puntos de corte de Cas9, pero la proteína aún podía encontrar su objetivo de ADN. También lograron combinar el tracrRNA y el ARN espaciador en una sola molécula llamada single-guide RNA (ARN guía único). Cuando esta molécula se une a Cas9, puede encontrar y cortar el ADN deseado. Se propuso que estos ARN guía sintéticos podrían usarse para la ingeniería genética.
La primera vez que se demostró que CRISPR funcionaba como una herramienta de ingeniería genética en células humanas fue en 2012. Desde entonces, se ha utilizado en muchos organismos, como la levadura del pan (Saccharomyces cerevisiae), el pez cebra (Danio rerio), la mosca de la fruta (Drosophila melanogaster), nematodos (Caenorhabditis elegans), plantas y ratones.
Además, CRISPR ha sido modificado para activar o desactivar ciertos genes.
Existen ahora muchas bibliotecas con miles de ARN guía diferentes.
La primera prueba de que CRISPR podía mejorar los síntomas de enfermedades en organismos vivos se dio en marzo de 2014. Investigadores del MIT lograron mejorar a ratones con desórdenes genéticos del hígado.
Otras proteínas Cas: Cas12a y CasΦ
En 2015, se descubrió otra nucleasa llamada Cpf1 (ahora conocida como Cas12a) en el sistema CRISPR/Cpf1 de la bacteria Francisella novicida. Cas12a tiene algunas diferencias importantes con Cas9:
- Corta el ADN de doble hebra de una manera diferente, dejando un corte "escalonado" en lugar de un corte "brusco".
- Necesita un tipo diferente de secuencia de reconocimiento (llamada PAM) que es rica en T.
- Solo necesita un tipo de ARN guía (CRRNA) para funcionar, mientras que Cas9 necesita dos.
Existen variantes de Cas12a, como AsCas12a y LbCas12a. Se ha encontrado una mutación en LbCas12a (llamada D156R) que mejora su eficiencia de corte en plantas y moscas de la fruta, siendo casi tan eficiente como Cas9.
En 2020, se describió una nueva nucleasa llamada CasΦ. Es mucho más pequeña que Cas9 o Cas12a, pero tiene características similares para la ingeniería genética. Se ha usado con éxito para modificar genes en moscas de la fruta de forma precisa, sin causar cortes no deseados en el ADN.
Herramientas anteriores para la edición de genes
Antes de CRISPR, los investigadores desarrollaron otras herramientas para la ingeniería genética:
- Las nucleasas con dedos de zinc (principios de los 2000): Eran proteínas que podían cortar el ADN en puntos específicos.
- Las nucleasas sintéticas llamadas TALENs: Eran más fáciles de usar que las de dedos de zinc.
Ambas técnicas requerían crear proteínas específicas para cada objetivo de ADN, lo cual era más complicado que usar los ARN guía de CRISPR. Los sistemas CRISPR son más eficientes y pueden llegar a más genes que estas técnicas anteriores.
Primer uso de CRISPR para "grabar" información
El 12 de julio de 2017, la revista británica Nature informó que investigadores de la Universidad de Harvard habían usado la técnica CRISPR para "grabar" la información digital de una fotografía y una animación corta (GIF) en el genoma de un grupo de bacterias. Luego, secuenciaron el ADN de los microorganismos para recuperar la imagen y el video con una precisión del 90%. Este avance abrió nuevas posibilidades en el campo de la "grabación molecular".
Cómo funciona CRISPR
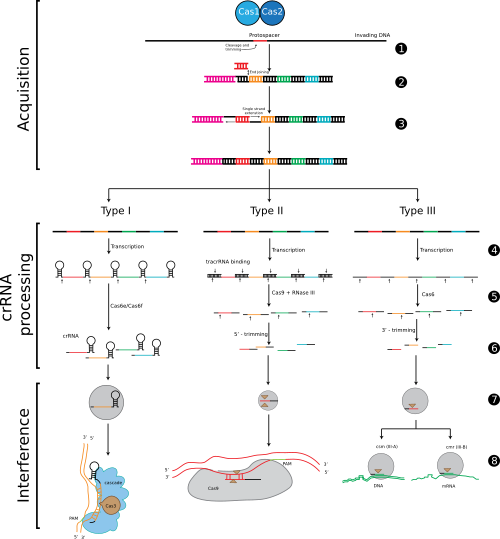
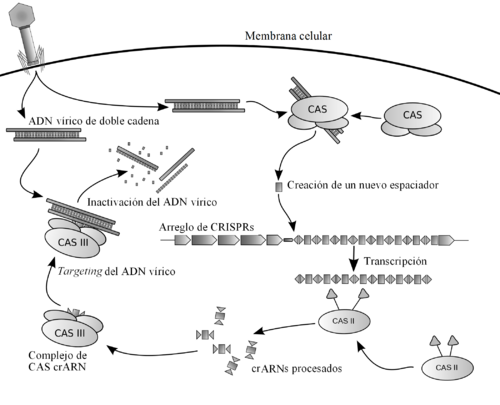
El sistema CRISPR funciona en varias etapas para defender a la bacteria de los virus:
Adquisición de los espaciadores
La primera etapa es cuando la bacteria "captura" un trozo del ADN del virus invasor y lo integra en su propio ADN como un espaciador. Los genes Cas1 y Cas2 son clave en este proceso. Se cree que Cas1 y Cas2 cortan fragmentos del ADN del virus y los insertan en el lugar correcto dentro de las secuencias CRISPR de la bacteria.
Los científicos descubrieron que los trozos de ADN de virus que se guardan como espaciadores no se eligen al azar. Siempre están junto a unas secuencias cortas de ADN llamadas PAM (por sus siglas en inglés). Los PAMs son importantes para que los sistemas CRISPR tipo I y II puedan reconocer dónde cortar el ADN del virus.
Los nuevos espaciadores se añaden a las secuencias CRISPR de forma ordenada, generalmente cerca de una secuencia especial llamada "líder".
Etapa de interferencia
Una vez que la bacteria tiene los espaciadores, el sistema CRISPR actúa en dos pasos:
- Creación de ARNcr: Las secuencias CRISPR se copian en una molécula larga de ARN. Luego, esta molécula se corta en pedazos más pequeños llamados ARNcr. La forma en que se cortan estos ARNcr varía según el tipo de sistema CRISPR-Cas.
- Interferencia guiada por ARNcr: Los ARNcr se unen a las proteínas Cas para formar complejos que pueden reconocer y atacar el ADN de los virus. Cuando un ARNcr encuentra una secuencia de ADN de virus que coincide con su espaciador, las proteínas Cas cortan y desactivan el ADN del virus.
Es importante que el sistema CRISPR sepa distinguir entre el ADN del virus y el propio ADN de la bacteria. Los ARNcr contienen una parte de la secuencia repetida de CRISPR. Esta parte de la repetición es la que evita que el sistema ataque el propio ADN de la bacteria, actuando como una señal de "ADN propio".
Aplicaciones de la tecnología CRISPR
La tecnología CRISPR/Cas ha abierto muchas puertas en la biotecnología:
Edición de genes
CRISPR puede añadir o quitar pequeñas partes del ADN en lugares muy específicos. Se ha usado para modificar varios genes a la vez.
Desactivación reversible de genes (Knockout reversible)
Existen versiones de CRISPR, llamadas "CRISPRi", que pueden desactivar genes de forma temporal y reversible, sin cortarlos. Esto es útil para estudiar la función de un gen.
Activación de genes
La proteína Cas9 también se ha usado para activar genes específicos en células humanas. Esto se logra dirigiendo múltiples elementos CRISPR a diferentes lugares en la región que controla el gen. Se ha aplicado a genes relacionados con enfermedades, el desarrollo muscular y la producción de ciertas proteínas.
Uso en la agricultura
En 2014, un investigador chino, Gao Caixia, solicitó patentes para crear una variedad de trigo resistente a una enfermedad llamada oídio. Se eliminaron genes que normalmente debilitan las defensas del trigo contra esta enfermedad. Esta nueva variedad podría reducir la necesidad de usar fungicidas.
Estudio de insectos
La tecnología CRISPR/Cas9 es muy importante para estudiar la función de los genes en insectos. Por ejemplo, se ha usado en el gusano de la seda (Bombyx mori) para identificar genes relacionados con su crecimiento, su resistencia a enfermedades y a la temperatura.
Identificación de elementos funcionales en el genoma
CRISPR también se usa para encontrar qué partes del ADN no codificante (ADN que no produce proteínas) tienen funciones importantes. Esto ayuda a entender cómo se regulan los genes y cómo se relacionan con ciertas características o enfermedades. Por ejemplo, se ha usado para identificar regiones del ADN que influyen en la resistencia a ciertos medicamentos.
Evolución y diversidad de CRISPR
Los estudios en Streptococcus thermophilus mostraron cómo los sistemas CRISPR influyen en la evolución de los fagos y las bacterias. Para que un espaciador CRISPR funcione, debe coincidir perfectamente con la secuencia del gen del fago. Si el fago muta (cambia) en esa secuencia, puede escapar de la defensa de la bacteria. Esto lleva a una "carrera armamentista" evolutiva, donde las bacterias adquieren nuevos espaciadores y los fagos desarrollan nuevas mutaciones.
La diversidad de CRISPR se ha estudiado en diferentes comunidades de bacterias, como las que se encuentran en los drenajes ácidos de minas o en la boca humana. Estos estudios muestran que los sistemas CRISPR evolucionan a diferentes velocidades y que la presencia de espaciadores varía mucho entre distintas poblaciones de bacterias.
Identificación de CRISPR en el ADN
Los sistemas CRISPR son fáciles de identificar en secuencias grandes de ADN porque tienen repeticiones y espaciadores muy característicos. Hay programas informáticos especiales como CRT, PILER-CR y CRISPRfinder que ayudan a los científicos a encontrar estas secuencias.
Analizar CRISPR en datos de metagenómica (el estudio de todo el ADN de una comunidad de microorganismos) es más difícil. Sin embargo, se están desarrollando métodos para extraer y reconstruir las secuencias CRISPR a partir de estos datos.
Importancia evolutiva
Los estudios muestran que los sistemas CRISPR se han conservado a lo largo de la evolución y se agrupan en tipos relacionados.
Gracias al mecanismo CRISPR/Cas, las bacterias pueden desarrollar inmunidad a ciertos virus, lo que detiene la propagación de esos virus. Por esta razón, se dice que los CRISPR/Cas son un mecanismo de herencia Lamarckiano, lo que significa que las bacterias pueden pasar a sus "hijos" una característica adquirida durante su vida (la inmunidad a un virus).
Las proteínas Cas9 son muy comunes en bacterias que causan enfermedades o que viven en nuestro cuerpo. La regulación por CRISPR/Cas puede ayudar a controlar los genes de las bacterias, especialmente en cómo interactúan con los organismos más grandes.
Automatización y apoyo a la investigación
Existe software gratuito para diseñar los ARN guía y encontrar cualquier gen deseado. Organizaciones como Addgene ofrecen a los investigadores la posibilidad de obtener componentes para su propio sistema CRISPR. En 2013, Addgene distribuyó más de 10,000 de estos componentes, lo que demuestra el gran interés en esta tecnología.
Propiedad intelectual
Los derechos sobre la patente de CRISPR han sido objeto de disputas.
En 2012, el equipo de Doudna solicitó una patente provisional para el uso del sistema CRISPR en la edición de genes. Sin embargo, Feng Zhang del Broad Institute también obtuvo una patente para el uso de CRISPR en células con núcleo (como las de animales y plantas, incluyendo las humanas). Esto generó controversia, ya que algunos científicos argumentaban que era lógico que la tecnología CRISPR funcionara en células humanas.
En 2017, la Oficina de Patentes de Estados Unidos dictaminó que las patentes del Broad Institute, que cubrían la aplicación de CRISPR/Cas9 en células eucariotas, eran diferentes de los inventos reclamados por la Universidad de California.
Varias empresas han obtenido licencias para usar la tecnología CRISPR en sus investigaciones y productos, como la creación de ratas y ratones genéticamente modificados para estudios.
Es importante recordar que el sistema CRISPR fue descubierto por primera vez por científicos japoneses en 1987, y luego redescubierto de forma independiente por el microbiólogo Francis Mojica en España.
Riesgos y consideraciones
Según investigadores del Instituto Wellcome Sanger en Inglaterra, la edición genética puede causar mutaciones no deseadas en lugares del genoma diferentes al sitio que se quería editar. La forma en que los genes interactúan entre sí es muy compleja, y los cambios no intencionados podrían afectar la salud de las personas con genes editados. Estas mutaciones, al ser genéticas, podrían ser transmitidas a la descendencia.
Es fundamental que la investigación en CRISPR continúe con mucha precaución para asegurar que la tecnología sea segura y precisa. Se están desarrollando herramientas para detectar estas mutaciones no deseadas, como el sistema VIVO, que permite comprobar si se producen cortes fuera de la región deseada.
Véase también
 En inglés: CRISPR Facts for Kids
En inglés: CRISPR Facts for Kids
- Edición génica CRISPR
- Nucleasas con dedos de zinc
- ADN no codificante
- CRISPR/Cpf1
- Edición de calidad
- Cas9
- Motivo adyacente de protoespaciador