Secuenciación del ADN para niños
La secuenciación del ADN es un conjunto de métodos y técnicas de bioquímica que sirven para descubrir el orden exacto de los nucleótidos en una cadena de ADN. Los nucleótidos son las "letras" de nuestro código genético: adenina (A), citosina (C), guanina (G) y timina (T). Conocer este orden es como leer un manual de instrucciones que tienen todos los seres vivos, desde las bacterias hasta las plantas y los animales.
Saber la secuencia del ADN es muy útil. Nos ayuda a entender cómo funcionan los seres vivos y cómo se desarrollan. También se usa en campos como la ciencia forense y para detectar cambios en el ADN que pueden estar relacionados con algunas enfermedades. Por ejemplo, se puede usar para identificar posibles problemas genéticos o para estudiar enfermedades raras.
El desarrollo de nuevas formas de secuenciar el ADN ha hecho que este proceso sea mucho más rápido. Esto fue clave para proyectos muy grandes como el Proyecto Genoma Humano, que buscaba leer todo el ADN de una persona. Gracias a estas técnicas, hoy conocemos el ADN completo de muchos animales, plantas y microorganismos.
Contenido
¿Cómo empezó la secuenciación del ADN?
Durante muchos años, la forma principal de secuenciar el ADN fue un método creado por Frederick Sanger en 1975. Antes de esto, a principios de los años 70, otros científicos como Walter Gilbert y Allan Maxam también desarrollaron sus propios métodos.
Curiosamente, la secuenciación del ARN (una molécula parecida al ADN) se desarrolló antes que la del ADN. Un gran logro fue cuando Walter Fiers y su equipo descubrieron la secuencia completa del genoma de un virus llamado Bacteriófago MS2.
El método de Maxam-Gilbert
En 1976-1977, Allan Maxam y Walter Gilbert crearon un método que usaba químicos para cortar el ADN en lugares específicos. Aunque fue importante, este método era complicado, usaba químicos peligrosos y no era fácil de usar a gran escala. Por eso, con el tiempo, fue reemplazado por otros métodos más sencillos y seguros.
Este método necesitaba marcar el ADN con radioisótopos y luego separarlo en un gel para ver los fragmentos.
Métodos de terminación de la cadena (Sanger)
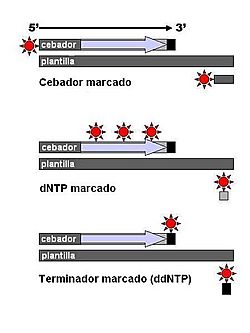
El método de terminación de la cadena, o método de Sanger, se volvió muy popular porque era más eficiente y seguro que el de Maxam-Gilbert. Usaba menos químicos peligrosos y menos radioactividad. La clave de este método es usar unas moléculas especiales llamadas didesoxinucleótidos (ddNTPs) que detienen la copia de la cadena de ADN.
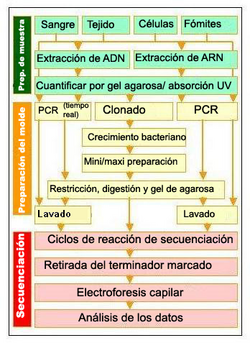
Para usar el método de Sanger, se necesita una cadena de ADN que sirva de molde, un "cebador" (un pequeño trozo de ADN que inicia la copia), una enzima llamada ADN polimerasa y los cuatro tipos de nucleótidos. Se añaden también los didesoxinucleótidos, que son como "letras" modificadas que, al unirse a la cadena, la detienen. Esto crea muchos fragmentos de ADN de diferentes tamaños.
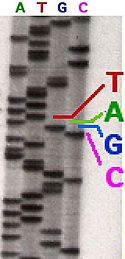
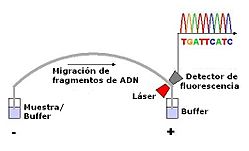
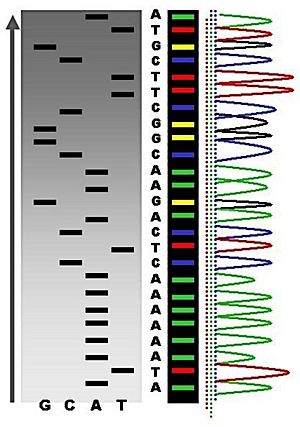
Luego, estos fragmentos se separan por tamaño usando una técnica llamada electroforesis en gel. Cada fragmento se visualiza, y al leer las bandas en el gel, se puede saber la secuencia del ADN.
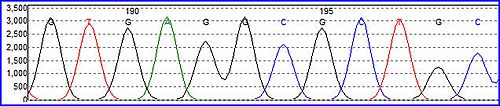
Existen variaciones de este método. Por ejemplo, se pueden usar marcadores fluorescentes en lugar de radiactivos. Esto permite que el proceso sea más rápido y se pueda automatizar con máquinas.
Secuenciación por terminador fluorescente
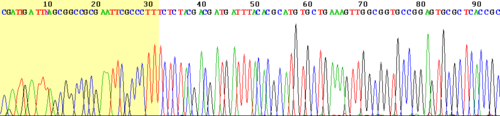
Una mejora importante fue marcar los didesoxinucleótidos (los que detienen la cadena) con diferentes colores fluorescentes. Así, cada "letra" (A, C, G, T) tiene un color distinto. La ventaja es que toda la secuenciación se puede hacer en una sola reacción, no en cuatro separadas.
Este método es muy usado hoy en día en máquinas automáticas, ya que permite leer muchas secuencias a la vez y de forma rápida.
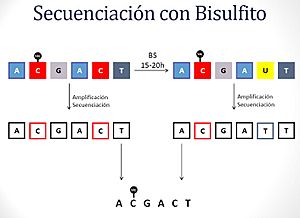
Secuenciación alelo-específica por bisulfito
Esta es una variación del método de Sanger que se usa para estudiar unas marcas especiales en el ADN llamadas "metilaciones". Estas metilaciones son como pequeños "post-it" que se pegan al ADN y pueden cambiar cómo funcionan los genes.
El método usa un químico llamado bisulfito que cambia algunas "letras" C (citosinas) que no tienen esa marca de metilación. Al comparar el ADN tratado con bisulfito con el ADN sin tratar, se puede saber dónde estaban esas marcas.
Pirosecuenciación
La pirosecuenciación es otro método para leer el ADN que no necesita geles ni marcadores fluorescentes. Se basa en detectar la luz que se produce cuando se añaden los nucleótidos a una cadena de ADN que se está copiando.
Funciona así: se añade un tipo de nucleótido a la vez. Si ese nucleótido es el correcto para la secuencia, se une y se libera una pequeña molécula que, a través de una serie de reacciones con otras enzimas, produce luz. Esta luz es detectada por una cámara. Si el nucleótido no es el correcto, se elimina y no hay luz.
La cantidad de luz que se emite nos dice cuántos nucleótidos iguales se han unido seguidos. Este método es bueno para leer fragmentos cortos de ADN y detectar cambios pequeños en la secuencia.
Automatización y preparación de las muestras
Las máquinas modernas de secuenciación de ADN pueden procesar cientos de muestras a la vez, trabajando muy rápido. Estas máquinas separan el ADN por tamaño y detectan los colores fluorescentes, mostrando los resultados en gráficos. Aunque las reacciones de secuenciación se hacen aparte, estas máquinas han hecho el proceso mucho más eficiente.
Estrategias de secuenciación a gran escala
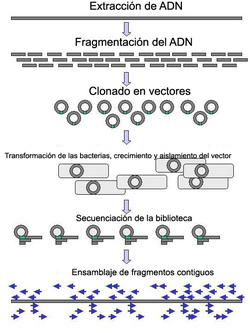
Los métodos actuales solo pueden leer fragmentos de ADN relativamente cortos (entre 300 y 1000 nucleótidos) en una sola vez. Para secuenciar genomas muy grandes, como el humano (que tiene miles de millones de "letras"), se necesita una estrategia especial.
Lo que se hace es cortar el ADN grande en muchos trozos pequeños. Estos trozos se copian muchas veces y luego se secuencian individualmente. Después, usando programas de computadora, se juntan todos esos trozos pequeños como si fueran piezas de un rompecabezas, buscando las partes que se superponen. Este proceso se llama "ensamblaje".
Este método es muy útil para secuenciar genomas completos por primera vez. Sin embargo, puede ser complicado si el ADN tiene muchas partes repetidas.
Cuando ya se conoce una secuencia de ADN y se quiere ver si hay algún cambio en ella, se usa la "resecuenciación". Se amplifica la parte de interés del ADN y se compara con la secuencia original para encontrar diferencias.
Nuevos métodos de secuenciación (Next-Generation Sequencing - NGS)
La necesidad de secuenciar ADN de forma más barata y rápida llevó al desarrollo de las tecnologías de "secuenciación de alto rendimiento" o "next-generation sequencing" (NGS). Estas tecnologías pueden leer millones de secuencias de ADN al mismo tiempo, lo que ha reducido mucho los costos y el tiempo.
NGS ha sido muy importante para la medicina, permitiendo entender mejor cómo los genes influyen en la salud y en las enfermedades.
Algunos de estos métodos de NGS son:
- Illumina (Solexa)
Este método usa polimerasas especiales y nucleótidos con marcadores fluorescentes que se pueden quitar. El ADN se pega a una superficie y se copia. Cada vez que se añade un nucleótido, se detiene la copia, se toma una foto para ver el color (la "letra") y luego se quita el marcador para que la copia continúe. Esto permite leer la secuencia paso a paso.
- Ion Torrent semiconductor
Este método detecta los iones de hidrógeno que se liberan cuando un nucleótido se une a la cadena de ADN. Si el nucleótido es el correcto, se produce una señal eléctrica que es detectada por un sensor. Es un método rápido y más económico porque no necesita marcadores fluorescentes.
- Secuenciación por ligación
Este método usa una enzima diferente, la ADN ligasa, para identificar la secuencia. Se basa en que esta enzima une pequeños trozos de ADN (oligonucleótidos) de forma específica, y esa unión produce una señal que revela la secuencia.
- Secuenciación de molécula única mediante PacBio
Esta tecnología permite leer fragmentos de ADN mucho más largos que otros métodos. Es muy útil para estudiar genomas complejos y detectar cambios grandes en el ADN. Una de sus ventajas es que puede identificar modificaciones en el ADN, como la metilación, directamente mientras lee la secuencia.
Secuenciación de una única molécula de ADN
Estos métodos son aún más avanzados y pueden secuenciar una sola molécula de ADN sin necesidad de hacer muchas copias antes.
El método tSMS de Helicos, por ejemplo, corta el ADN en fragmentos pequeños y los pega a una superficie. Luego, va añadiendo nucleótidos marcados con fluorescencia uno por uno. Una cámara toma fotos para ver qué nucleótido se ha unido en cada posición. Al final, un programa de computadora junta todas las piezas para reconstruir la secuencia completa.
Otro método es la secuenciación SMRT de Pacific Biosciences (PacBio). En este caso, la enzima que copia el ADN está fija en pequeños agujeros. Los nucleótidos tienen marcadores fluorescentes en una parte que se libera cuando se unen a la cadena. Así, la luz se detecta justo en el momento de la unión, haciendo el proceso muy rápido.
RIMS-seq2
RIMS-seq2 es una técnica nueva que no solo secuencia el ADN, sino que también puede ver las marcas de metilación al mismo tiempo. Esto es importante porque la metilación del ADN es un proceso biológico fundamental que influye en cómo funcionan nuestros genes. Esta técnica es más sencilla y económica que otras para obtener ambas informaciones a la vez.
Otras tecnologías de secuenciación
Existen otras ideas y métodos en desarrollo. Algunos usan chips de ADN para ver cómo el ADN desconocido se une a secuencias conocidas. Otros usan la espectrometría de masas para medir el peso de los fragmentos de ADN.
También se están investigando métodos que leen la secuencia de ADN a medida que la cadena pasa por poros muy pequeños (nanoporos). Otros usan microscopios muy potentes para ver los nucleótidos directamente.
Principales avances en la secuenciación del ADN
- 1953: Se descubre la estructura de la doble hélice de ADN.
- 1972: Se desarrolla la tecnología del ADN recombinante, que permite aislar trozos específicos de ADN.
- 1975: Se secuencia por primera vez el genoma completo de un virus de ADN (el bacteriófago φX174).
- 1977: Allan Maxam y Walter Gilbert publican su método de secuenciación química. Fred Sanger publica su método de secuenciación enzimática.
- 1980: Frederick Sanger y Walter Gilbert ganan el Premio Nobel de química.
- 1982: Se crea GenBank, una base de datos pública para guardar secuencias de ADN.
- 1983: Kary Mullis y su equipo desarrollan la reacción en cadena de la polimerasa (PCR), una técnica para copiar pequeños fragmentos de ADN.
- 1987: Applied Biosystems lanza la primera máquina automática de secuenciación.
- 1990: El NIH de Estados Unidos comienza grandes proyectos de secuenciación de varios organismos.
- 1995: Se publica la primera secuencia de un organismo que vive libremente, la bacteria Haemophilus influenzae.
- 1998: Se funda Celera Genomics con el objetivo de secuenciar el genoma humano en 3 años.
- 2000: Se anuncian los primeros borradores de la secuencia del genoma humano.
- 2001: El Proyecto Genoma Humano publica el borrador de la secuencia del genoma humano.
- 2007: Craig Venter publica su genoma humano completo, el primero en ser secuenciado por completo.
Véase también
 En inglés: Sequence analysis Facts for Kids
En inglés: Sequence analysis Facts for Kids
- Análisis moleculares de ADN
- Secuenciación 454
- Proyecto Genoma Humano
- Medicina genómica
- Metagenómica
- Transistor de ADN de efecto de campo
- Bioinformática