Bioinformática para niños
La bioinformática es un campo de estudio que combina la Biología con la informática y la estadística. Imagina que tienes muchísimos datos sobre seres vivos, como la información genética de una planta o de un animal. La bioinformática usa computadoras y programas especiales para organizar, analizar y entender toda esa información.
A veces, escucharás términos como biología computacional o informática biológica. Aunque son muy parecidos y a menudo se usan como sinónimos, cada uno tiene un enfoque ligeramente diferente. La bioinformática se centra más en crear las herramientas y programas para manejar los datos biológicos. La biología computacional, en cambio, se dedica a usar esas herramientas para investigar y entender cómo funcionan los sistemas biológicos, como las células o los organismos completos.
Los científicos que trabajan en bioinformática usan conocimientos de matemática aplicada, estadística, ciencias de la computación, inteligencia artificial y química. Esto les ayuda a resolver problemas muy grandes que serían imposibles de manejar solo con la mente humana. Por ejemplo, pueden analizar el ADN de miles de organismos o simular cómo interactúan las proteínas.
Algunos de los trabajos más importantes en bioinformática incluyen:
- Comparar secuencias de ADN y proteínas.
- Encontrar genes en el genoma.
- Predecir la forma tridimensional de las proteínas.
- Entender cómo se expresan los genes.
- Estudiar cómo evolucionan los seres vivos.
Una de las tareas principales es extraer información útil de los datos que se obtienen con técnicas biológicas avanzadas, como la secuenciación del genoma. Por ejemplo, armar el genoma completo de un organismo a partir de pequeños fragmentos de ADN es un gran desafío que la bioinformática ayuda a resolver.
Contenido
- ¿Qué es la Bioinformática?
- Breve Historia de la Bioinformática
- Áreas Clave de Investigación
- Análisis de Secuencias
- Anotación de Genomas
- Biología Evolutiva Computacional
- Medición de la Biodiversidad
- Análisis de la Expresión Génica
- Análisis de la Regulación Génica
- Análisis de la Expresión de Proteínas
- Análisis de Mutaciones en el Cáncer
- Predicción de la Estructura de las Proteínas
- Genómica Comparativa
- Modelado de Sistemas Biológicos
- Análisis de Imagen de Alto Rendimiento
- Acoplamiento Proteína-Proteína
- Ontologías e Integración de Datos
- Herramientas de Software
- Véase también
¿Qué es la Bioinformática?
Como ya mencionamos, la bioinformática, la biología computacional y la biocomputación son términos que se usan mucho. Sin embargo, algunas instituciones importantes, como los Institutos Nacionales de la Salud de Estados Unidos (NIH), los definen de forma más específica:
- Bioinformática: Se trata de investigar, desarrollar y usar herramientas y métodos computacionales para aprovechar al máximo los datos biológicos, médicos o de salud. Esto incluye herramientas para obtener, guardar, organizar, analizar o mostrar esos datos.
- Biología computacional: Es el desarrollo y la aplicación de métodos teóricos, análisis de datos, modelos matemáticos y simulaciones por computadora para estudiar sistemas biológicos, como las células o los organismos.
En resumen, la bioinformática se enfoca más en la información y las herramientas para manejarla, mientras que la biología computacional se centra en las hipótesis y en cómo usar los datos para entender la biología.
El término biocomputación se refiere a la creación y uso de computadoras que tienen componentes biológicos o que funcionan de manera similar a los seres vivos. Es un campo más experimental y futurista.
Breve Historia de la Bioinformática
La bioinformática es un campo relativamente nuevo, pero ha crecido muy rápido gracias a los avances en la biología y la tecnología de las computadoras.
Los Comienzos: Años 50, 60 y 70
En los años 50, hubo descubrimientos clave en biología, como la estructura de doble hélice del ADN propuesta por Watson y Crick en 1953. También se secuenció la primera proteína (la insulina bovina) en 1955. Al mismo tiempo, la tecnología de computadoras avanzaba, con la creación del primer circuito integrado en 1958.
En los años 60, Margaret Dayhoff, una pionera de la bioinformática, publicó el primer "Atlas de Secuencias de Proteínas" (1965). Este fue un trabajo fundamental que sentó las bases para las bases de datos de proteínas que usamos hoy. También en esta década, se desarrollaron los protocolos para conectar computadoras en red, lo que llevó al nacimiento de ARPANET en 1969, el precursor de Internet.
Los años 70 vieron el desarrollo de algoritmos importantes, como el algoritmo Needleman-Wunsch (1970) para comparar secuencias. Se creó el primer banco de datos de proteínas (Protein Data Bank, 1971) y se logró secuenciar el ADN por primera vez en 1977. En 1978, se publicó la secuencia completa de genes de un organismo, el fago Φ-X174.
Años 80: Grandes Avances
La década de los 80 fue muy importante para la bioinformática:
- Descubrimientos científicos: En 1982, se secuenció el genoma del fago lambda usando una nueva técnica llamada secuenciación "shotgun" (por perdigonada). También se descubrió la PCR (1983), una técnica que permite multiplicar muestras de ADN para analizarlas mejor.
- Herramientas bioinformáticas: Aparecieron algoritmos clave como el algoritmo Smith-Waterman (1981) y FASTA (1988) para comparar secuencias. Se crearon bases de datos biológicas muy importantes como GenBank (1982) y Swiss-Prot (1986). Además, se empezó a planear el Proyecto Genoma Humano en 1985, un esfuerzo gigantesco para secuenciar todo el genoma humano.
- Avances tecnológicos: Surgieron lenguajes de programación útiles para la bioinformática, como PERL (1987).
Años 90: La Era de Internet y los Genomas
En los años 90, la secuenciación de genomas avanzó rápidamente. En 1995, se secuenciaron los primeros genomas completos de bacterias, y en 1996, el primer genoma eucariota (el de la levadura). Al final de la década, se secuenció el primer cromosoma humano completo (el cromosoma 22).
En bioinformática, se desarrolló BLAST (1990), una herramienta muy popular para buscar similitudes entre secuencias. También apareció ClustalW (1994) para alinear múltiples secuencias. Un hito tecnológico crucial fue la invención de la World Wide Web por Tim Berners-Lee en 1990, que facilitó enormemente el intercambio de información científica.
Siglo XXI: Genomas Completos y Más
Los primeros años del siglo XXI vieron la culminación de grandes proyectos. En 2001, se publicó el borrador del genoma humano, y en 2003, el Proyecto Genoma Humano se completó antes de lo previsto. Desde entonces, se han secuenciado los genomas de muchísimos organismos, desde plantas hasta animales.
La bioinformática sigue evolucionando, con programas cada vez más potentes y la creación de institutos especializados en todo el mundo.
Áreas Clave de Investigación
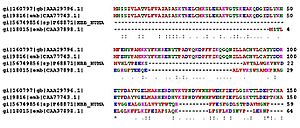
Análisis de Secuencias
Desde que se secuenció el primer organismo, se han decodificado y guardado en bases de datos las secuencias de ADN de cientos de miles de organismos. Los programas de computadora son esenciales para analizar estos datos, ya que hacerlo a mano sería imposible. Estos programas pueden encontrar genes, identificar secuencias reguladoras y comparar genes entre diferentes especies para ver sus similitudes y relaciones evolutivas.
La secuenciación "shotgun" es una técnica que produce miles de pequeños fragmentos de ADN. La bioinformática se encarga de "ensamblar" estos fragmentos, como si fuera un rompecabezas gigante, para reconstruir el genoma completo del organismo. Este proceso es muy complejo, especialmente para genomas grandes como el humano.
La bioinformática también ayuda a encontrar genes y secuencias reguladoras dentro de un genoma. No todo el ADN codifica proteínas; hay grandes partes que no tienen una función obvia, pero que pueden contener elementos importantes aún por descubrir.

Anotación de Genomas
Cuando se secuencia un genoma, la anotación es el proceso de marcar los genes y otras características biológicas en la secuencia de ADN. El primer sistema de software para esto fue creado en 1995. Hoy en día, los programas de anotación genómica son mucho más avanzados y están en constante mejora.
Biología Evolutiva Computacional
La Biología evolutiva estudia cómo las especies cambian y se originan a lo largo del tiempo. La informática ha sido de gran ayuda en este campo:
- Permite seguir la evolución de muchos organismos analizando los cambios en su ADN.
- Permite comparar genomas completos para entender eventos evolutivos complejos, como la duplicación de genes.
- Ayuda a construir modelos computacionales de poblaciones para predecir cómo cambiarán con el tiempo.
- Facilita el seguimiento y la compartición de información sobre un número creciente de especies.
El objetivo futuro es reconstruir el complejo árbol filogenético de la vida, que muestra las relaciones de parentesco entre todas las especies.
Medición de la Biodiversidad
La biodiversidad se refiere a la variedad de vida en un ecosistema. La bioinformática utiliza bases de datos para recopilar información sobre especies, sus descripciones, dónde viven, su información genética, el tamaño de sus poblaciones y cómo interactúan con otras especies.
Se usa software especializado para encontrar, visualizar y analizar esta información, y lo más importante, para compartirla. Las simulaciones por computadora pueden modelar la dinámica de las poblaciones o calcular cómo mejorar la diversidad genética de una especie en peligro. Un aspecto emocionante es la posibilidad de guardar las secuencias completas de ADN de especies amenazadas, lo que permitiría estudiar su genética incluso si se extinguieran.
Análisis de la Expresión Génica
La expresión génica es el proceso por el cual la información de un gen se usa para crear un producto funcional, como una proteína. La bioinformática ayuda a medir los niveles de ARNm (una molécula que lleva la información del ADN para hacer proteínas) usando técnicas como los microarrays de ADN.
Estas técnicas pueden generar muchos datos, y la bioinformática desarrolla herramientas estadísticas para separar la información importante del "ruido" (datos irrelevantes). Estos estudios se usan a menudo para identificar los genes involucrados en enfermedades, como el Cáncer.
Análisis de la Regulación Génica
La regulación génica es el control de cuándo y cómo se activan o desactivan los genes. La bioinformática explora este proceso en varios pasos. Por ejemplo, analiza las regiones del ADN llamadas promotores, que influyen en la transcripción de un gen.
Los datos de expresión génica se pueden usar para entender la regulación. Al comparar datos de diferentes estados de un organismo (por ejemplo, células sanas y células enfermas), se pueden formular hipótesis sobre qué genes están involucrados en cada estado.
Análisis de la Expresión de Proteínas
La bioinformática también apoya el análisis de proteínas. Técnicas como los microarrays de proteínas y la espectrometría de masas de alto rendimiento permiten obtener una "instantánea" de las proteínas presentes en una muestra biológica. La bioinformática ayuda a procesar y analizar estas grandes cantidades de datos para identificar y estudiar las proteínas.
Análisis de Mutaciones en el Cáncer
En el cáncer, el genoma de las células afectadas cambia de maneras complejas. Se están realizando grandes esfuerzos de secuenciación para identificar cambios en el ADN de las células cancerosas. Los bioinformáticos crean sistemas automatizados y nuevos algoritmos para manejar el enorme volumen de datos y comparar las secuencias de ADN de tumores con el genoma humano normal.
Estos análisis ayudan a identificar las mutaciones que contribuyen al desarrollo del cáncer, lo que es crucial para el diagnóstico y el desarrollo de nuevos tratamientos.
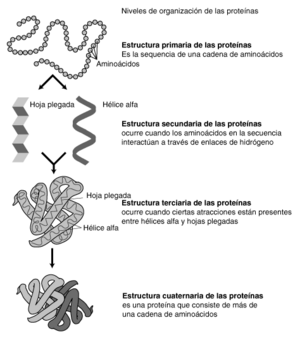
Predicción de la Estructura de las Proteínas
La predicción de la estructura de las proteínas es una aplicación muy importante de la bioinformática. La secuencia de aminoácidos de una proteína (su estructura primaria) determina su forma tridimensional. Conocer esta forma es vital para entender cómo funciona la proteína.
La bioinformática utiliza el concepto de homología: si dos proteínas tienen secuencias similares, es probable que también tengan estructuras y funciones parecidas. Esta idea se usa en el "modelado por homología" para predecir la estructura de una proteína si ya se conoce la estructura de una proteína similar.
Genómica Comparativa
La Genómica comparativa consiste en comparar los genomas de diferentes organismos para entender cómo han evolucionado. Esto permite rastrear los procesos evolutivos que causan las diferencias entre dos genomas, desde pequeños cambios en nucleótidos individuales hasta grandes reordenamientos cromosómicos.
Este campo plantea muchos desafíos para los desarrolladores de modelos matemáticos y algoritmos, ya que la evolución del genoma es muy compleja.
Modelado de Sistemas Biológicos
La biología de sistemas utiliza simulaciones por computadora de partes de las células (como las redes de metabolismo o las redes de regulación genética) para analizar y visualizar las complejas conexiones de estos procesos. También se usa para entender los procesos evolutivos mediante la simulación de formas de vida artificiales.
Análisis de Imagen de Alto Rendimiento
Las tecnologías computacionales se usan para acelerar y automatizar el procesamiento y análisis de grandes cantidades de imágenes biomédicas. Estos sistemas mejoran la precisión y la rapidez del análisis, y son cada vez más importantes para el diagnóstico y la investigación.
Algunos ejemplos incluyen la cuantificación de células, el análisis de imágenes médicas y la determinación de patrones en el flujo de aire pulmonar.
Acoplamiento Proteína-Proteína
Se han determinado las estructuras tridimensionales de miles de proteínas. Una pregunta clave es si se pueden predecir las posibles interacciones proteína-proteína basándose solo en sus formas 3D. Se han desarrollado varios métodos para abordar este problema, aunque todavía hay mucho trabajo por hacer.
Ontologías e Integración de Datos
Las ontologías biológicas son como diccionarios especiales que organizan conceptos y descripciones biológicas de una manera que las computadoras pueden entender y analizar fácilmente. Esto permite obtener un valor adicional al analizar la información de forma integrada.
Un ejemplo muy usado es la ontología génica, que describe la función de los genes.
Herramientas de Software
La bioinformática utiliza una gran variedad de programas de software, desde herramientas sencillas hasta programas gráficos complejos y servicios web.
Una de las herramientas más conocidas es BLAST, un algoritmo que busca similitudes entre secuencias de ADN o proteínas. El NCBI (Centro Nacional de Información Biotecnológica de EE. UU.) ofrece una versión de BLAST muy utilizada en línea.
Para alinear múltiples secuencias, el software de referencia es ClustalW. También hay muchos otros programas para diferentes objetivos, como la predicción de estructuras de proteínas o el modelado de sistemas biológicos.
Software Libre en Bioinformática
Mucho software de bioinformática es de software libre, lo que significa que su código es accesible y puede ser modificado y distribuido libremente. Esto ha permitido que muchos grupos de investigación contribuyan al campo, sin importar sus fuentes de financiación. El software libre a menudo sirve como un lugar para probar nuevas ideas y establecer estándares.
Algunos ejemplos de software libre en bioinformática son Bioconductor, BioPerl, Biopython y EMBOSS. La organización sin fines de lucro Open Bioinformatics Foundation patrocina una conferencia anual sobre software libre en bioinformática.
Servicios Web en Bioinformática
Los servicios web permiten que una aplicación en una computadora use algoritmos, datos y recursos de computación alojados en servidores en cualquier parte del mundo. Esto es muy útil porque los usuarios no tienen que preocuparse por actualizar el software o las bases de datos.
Estos servicios web se clasifican en categorías como:
- Búsquedas de similitudes entre secuencias (como BLAST).
- Alineamientos múltiples de secuencias (como ClustalW).
- Análisis estructural.
- Acceso a bases de datos y literatura especializada.
Sistemas de Gestión de Flujo de Trabajo de Bioinformática
Un sistema de gestión de flujo de trabajo de bioinformática es un programa diseñado para ayudar a los científicos a organizar y ejecutar una serie de pasos computacionales o de manipulación de datos. Estos sistemas facilitan la creación, ejecución, compartición y reutilización de flujos de trabajo, y permiten rastrear el origen de los resultados.
Algunas plataformas que ofrecen este servicio son Galaxy, Kepler y Taverna.
BioCompute Objects (BCO)
Los "BioCompute Objects" son como "cuadernos de laboratorio digitales" que permiten que los protocolos de bioinformática sean reproducibles, se puedan replicar, revisar y reutilizar. Esto es muy importante para que los científicos puedan compartir sus métodos y resultados de forma transparente y para que las agencias reguladoras puedan entender cómo se obtuvieron los datos.
Véase también
 En inglés: Bioinformatics Facts for Kids
En inglés: Bioinformatics Facts for Kids