Factor de transcripción para niños
En el mundo de la biología molecular y la genética, un factor de transcripción es una proteína muy especial. Su trabajo principal es unirse a partes específicas del ADN para controlar cómo se copia la información genética del ADN a un tipo de ARN llamado ARN mensajero. Piensa en ellos como los directores de orquesta de nuestros genes.
Estos factores pueden trabajar solos o con otras proteínas. Su función es ayudar a que la ARN polimerasa (la enzima que hace las copias del ADN) se una a ciertos genes. Así, pueden "encender" (como un activador) o "apagar" (como un represor) la copia de la información genética.
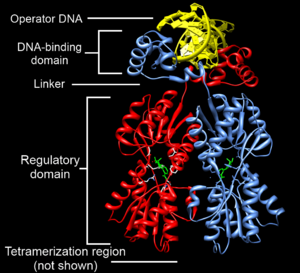
Una característica clave de los factores de transcripción es que tienen una parte especial llamada "dominio de unión al ADN". Esta parte es la que les permite pegarse a las secuencias específicas de ADN que están cerca de los genes que controlan. Otras proteínas también ayudan en la regulación de los genes, pero como no tienen este dominio de unión al ADN, no se consideran factores de transcripción.
Contenido
- ¿Por qué son tan importantes los factores de transcripción?
- ¿Cómo funcionan los factores de transcripción?
- Funciones clave de los factores de transcripción
- ¿Cómo se regulan los factores de transcripción?
- ¿Cómo están hechos los factores de transcripción?
- Importancia en la salud
- Papel en la evolución
- Tipos de factores de transcripción
- Ver también
- Véase también
¿Por qué son tan importantes los factores de transcripción?
Presentes en todos los seres vivos
Los factores de transcripción son esenciales para que los genes funcionen correctamente. Por eso, los encontramos en todos los seres vivos, desde las bacterias más pequeñas hasta los seres humanos. Cuanto más grande y complejo es un organismo, más factores de transcripción suele tener.
En el genoma humano, que es el conjunto completo de nuestro ADN, hay miles de proteínas que tienen dominios de unión al ADN. Se cree que muchas de ellas actúan como factores de transcripción. De hecho, ¡alrededor del 10% de nuestros genes producen factores de transcripción! Esto los convierte en una de las familias de proteínas más grandes de nuestro cuerpo.
Además, cada gen suele tener varios lugares donde diferentes factores de transcripción pueden unirse. Para que un gen funcione de manera eficiente, varios factores de transcripción deben trabajar juntos. Esta combinación de factores ayuda a que cada gen se regule de forma única durante el desarrollo de un organismo.
¿Cómo funcionan los factores de transcripción?
Los factores de transcripción se unen a zonas especiales del ADN llamadas "potenciadoras" o "promotoras", que están cerca de los genes que controlan. Dependiendo del factor, pueden aumentar o disminuir la copia de ese gen.
Usan diferentes maneras para regular la actividad de los genes:
- Pueden ayudar o impedir que la ARN polimerasa se una al ADN.
- Pueden modificar unas proteínas llamadas histonas, que son como carretes donde se enrolla el ADN.
- Algunas proteínas, como las histona acetiltransferasas (HAT), añaden una pequeña molécula (acetilo) a las histonas. Esto hace que el ADN se suelte un poco, facilitando que se copie el gen.
- Otras, como las histona deacetilasas (HDAC), quitan esa molécula, haciendo que el ADN se enrolle más fuerte y sea más difícil de copiar.
- También pueden reclutar otras proteínas que les ayuden en su trabajo.
Funciones clave de los factores de transcripción
Los factores de transcripción son como los lectores e intérpretes de los "planos" genéticos que están en nuestro ADN. Se unen al ADN y ayudan a iniciar o detener la copia de los genes. Por eso, son vitales para muchos procesos en nuestras células.
Control de la copia básica de genes
En organismos complejos como nosotros (eucariontes), hay un grupo importante de factores de transcripción llamados "factores generales de transcripción". Estos son necesarios para que cualquier gen pueda empezar a copiarse. Muchos de ellos no se unen directamente al ADN, sino que forman parte de un equipo más grande que interactúa con la ARN polimerasa.
Regulación específica de genes
Otros factores de transcripción controlan la actividad de genes específicos. Se unen a las regiones "potenciadoras" del ADN. Son cruciales para asegurar que los genes se activen en la célula correcta, en el momento adecuado y en la cantidad necesaria para el organismo.
Desarrollo de los seres vivos
Muchos factores de transcripción son fundamentales en el desarrollo de los organismos multicelulares. Responden a señales del ambiente y activan o desactivan los genes necesarios. Esto permite cambios en la forma de las células o en sus actividades, lo que es clave para que las células sepan en qué se convertirán. Por ejemplo, los factores de transcripción de los genes Hox son importantes para formar el patrón corporal en animales como las moscas o los humanos.
Comunicación entre células
Las células se comunican liberando moléculas que activan una cadena de señales en otras células. Si esta señal necesita cambiar la actividad de los genes, los factores de transcripción suelen ser los encargados de llevar a cabo esa acción. Un ejemplo es la señalización del estrógeno, una hormona que viaja al núcleo de la célula y se une a un factor de transcripción, cambiando la actividad de ciertos genes.
Respuesta al entorno
Los factores de transcripción también ayudan a las células a responder a cambios en el ambiente. Por ejemplo, el factor de choque térmico (HSF) activa genes que ayudan a las células a sobrevivir a altas temperaturas. El factor inducible por hipoxia (HIF) ayuda a las células a sobrevivir con poco oxígeno.
Control del ciclo celular
Muchos factores de transcripción, incluyendo algunos que pueden estar relacionados con el cáncer, ayudan a regular el ciclo celular. Deciden cuándo una célula debe dividirse en dos. Un ejemplo es el gen Myc, que es importante para el crecimiento celular y la muerte celular programada.
Papel en enfermedades
Los factores de transcripción también pueden ser usados por bacterias para alterar los genes de una célula huésped y así causar enfermedades. Por ejemplo, algunas bacterias inyectan proteínas en las plantas que activan genes que les ayudan a infectarlas.
¿Cómo se regulan los factores de transcripción?
Los procesos importantes en biología suelen tener muchos niveles de control, y los factores de transcripción no son la excepción. No solo controlan la actividad de otros genes, sino que su propia actividad también está regulada.
Producción
Los factores de transcripción, como todas las proteínas, se producen a partir de un gen. La cantidad de un factor de transcripción puede regularse en cualquier paso de este proceso. A veces, un factor de transcripción puede incluso regular su propia producción. Por ejemplo, si hay mucho de un factor, puede unirse a su propio gen para disminuir su producción, manteniendo así un nivel estable.
Ubicación en la célula
En las células eucariontes, los factores de transcripción se producen en el citoplasma pero actúan en el núcleo celular. Muchos tienen "señales de localización nuclear" que los dirigen al núcleo. Para algunos, esta es una parte clave de su regulación: solo pueden ir al núcleo después de unirse a una molécula específica en el citoplasma.
Activación
Los factores de transcripción pueden "encenderse" o "apagarse" de varias maneras:
- Unión a moléculas: Algunas moléculas pueden unirse a un factor de transcripción, no solo cambiando su ubicación, sino también activándolo para que pueda unirse al ADN.
- Fosforilación: Añadir un grupo fosfato a un factor de transcripción puede activarlo, permitiéndole unirse al ADN.
- Interacción con otras proteínas: Muchos factores de transcripción necesitan unirse a otros factores o proteínas para funcionar correctamente.
Acceso al ADN
En las células eucariontes, el ADN está muy enrollado alrededor de las histonas, formando estructuras compactas llamadas nucleosomas. Esto hace que el ADN sea difícil de alcanzar para muchos factores de transcripción. Algunos factores, llamados "factores pioneros", pueden unirse al ADN incluso cuando está enrollado. Para la mayoría, el ADN debe desenrollarse un poco para que puedan acceder a él.
Disponibilidad de ayudantes
La mayoría de los factores de transcripción no trabajan solos. Para que un gen se copie, varios factores de transcripción deben unirse a las secuencias reguladoras del ADN. Este grupo de factores recluta a otras proteínas que actúan como "cofactores", que son esenciales para que la ARN polimerasa se una eficientemente.
¿Cómo están hechos los factores de transcripción?
Los factores de transcripción tienen una estructura modular, lo que significa que están formados por diferentes partes o "dominios":
- Dominio de unión a ADN (DBD): Esta es la parte que se une a las secuencias específicas de ADN, como los potenciadores o promotores, que están cerca de los genes que regulan. Las secuencias de ADN a las que se unen se llaman "elementos de respuesta".
- Dominio de transactivación (TAD): Esta parte tiene sitios para que se unan otras proteínas que ayudan a regular la copia de los genes.
- Dominio de detección de señal (SSD): Esta parte es opcional y detecta señales del exterior. Luego, transmite estas señales al resto del equipo de transcripción, lo que lleva a que el gen se active o desactive. A veces, el DBD y el SSD pueden estar en proteínas diferentes que se unen para trabajar juntas.
Dominio de unión al ADN
La parte del factor de transcripción que se une al ADN se llama dominio de unión al ADN. Hay muchas familias diferentes de estos dominios, cada una con una forma particular de unirse al ADN. Algunos ejemplos incluyen las "hélices-giro-hélice" y los "dedos de zinc".
Elementos de respuesta
La secuencia de ADN a la que se une un factor de transcripción se conoce como "sitio de unión del factor de transcripción" o "elemento de respuesta". Los factores de transcripción se unen a estas secuencias usando fuerzas químicas. Aunque se unen a secuencias específicas, no todas las bases de ADN en el sitio de unión tienen que interactuar con el factor. Esto significa que un factor de transcripción puede unirse a un grupo de secuencias similares, cada una con una fuerza de unión diferente.
Por ejemplo, la proteína de unión a TATA (TBP) se une principalmente a la secuencia TATAAAA, pero también puede unirse a secuencias parecidas como TATATAT.
Como los factores de transcripción pueden unirse a secuencias similares y estas secuencias suelen ser cortas, es posible que aparezcan sitios de unión potenciales al azar en el ADN. Sin embargo, un factor de transcripción rara vez se une a todas las secuencias compatibles en el genoma de una célula. Otros factores, como la accesibilidad del ADN o la disponibilidad de cofactores, también influyen en dónde se unirá un factor de transcripción. Por eso, es difícil predecir con exactitud dónde se unirá un factor de transcripción en una célula viva solo conociendo la secuencia del ADN.
Importancia en la salud
Los factores de transcripción son muy importantes en la medicina por dos razones principales: (1) si hay cambios (mutaciones) en ellos, pueden causar enfermedades, y (2) pueden ser el objetivo de algunos medicamentos.
Enfermedades relacionadas
Debido a su papel crucial en el desarrollo, la comunicación entre células y el ciclo celular, algunas enfermedades humanas están relacionadas con mutaciones en los factores de transcripción.
Muchos factores de transcripción están relacionados con el cáncer. Si mutan o se regulan de forma anormal, pueden contribuir al desarrollo de esta enfermedad. Algunas familias importantes de factores de transcripción en el cáncer humano son NF-κB, AP-1 y STAT, así como los receptores esteroideos.
Otros ejemplos de enfermedades asociadas a mutaciones en factores de transcripción incluyen:
- Síndrome de Rett: Causado por mutaciones en el factor de transcripción MECP2.
- Diabetes (tipo MODY): Puede ser causada por mutaciones en factores nucleares de hepatocitos (HNFs) o el factor-1 del promotor de insulina (IPF1/Pdx1).
- Dispraxia verbal del desarrollo: Relacionada con mutaciones en el factor de transcripción FOXP2.
- Enfermedades autoinmunes (IPEX): Causadas por mutaciones en el factor de transcripción FOXP3.
- Síndrome de Li-Fraumeni: Causado por mutaciones en el supresor de tumores p53.
- Cáncer de mama: La familia STAT es relevante en este tipo de cáncer.
- Múltiples cánceres: La familia HOX está involucrada en varios tipos de cáncer.
Posibles objetivos para medicamentos
Alrededor del 10% de los medicamentos que se recetan hoy en día actúan sobre los receptores nucleares, que son un tipo de factores de transcripción. Ejemplos incluyen medicamentos para el cáncer de mama y próstata, así como algunos antiinflamatorios. Además, muchos medicamentos afectan indirectamente a los factores de transcripción a través de las cadenas de señales en las células. Se investiga si otros factores de transcripción podrían ser objetivos para nuevos tratamientos.
Papel en la evolución
La duplicación de genes ha sido muy importante para la evolución de las especies, y esto es especialmente cierto para los factores de transcripción. Cuando un gen se duplica, una de las copias puede cambiar sin afectar negativamente la función original. Esto permite que los factores de transcripción evolucionen y adquieran nuevas funciones, lo que ha sido clave en el desarrollo de la diversidad de la vida.
Tipos de factores de transcripción
Los factores de transcripción se pueden clasificar de varias maneras: por cómo funcionan, por su función reguladora o por la similitud de su estructura.
Clasificación por su mecanismo
Hay tres tipos principales según cómo actúan:
- Factores generales de la transcripción: Son necesarios para que el proceso de copia de genes comience. Interactúan con la zona inicial de los genes.
- Factores de transcripción específicos: Se unen a lugares específicos del ADN para estimular o detener la copia de un gen. Varían mucho dependiendo del gen que regulan.
Clasificación por su función
También se pueden clasificar según su función reguladora:
- Siempre activos: Están presentes en todas las células y siempre funcionando.
- Activos bajo ciertas condiciones: Necesitan una señal para activarse.
- Dependientes del desarrollo: Su expresión está muy controlada, pero una vez que se producen, no necesitan más activación.
- Dependientes de señales: Necesitan una señal externa para activarse.
- Señales de fuera de la célula (hormonas).
- Señales de dentro de la célula (pequeñas moléculas).
- Señales de la membrana celular (activan cadenas de señales que los modifican).
Clasificación por su estructura
Los factores de transcripción también se clasifican por la forma de sus dominios de unión al ADN. Hay muchas "superclases" y "clases" basadas en estas estructuras, como los dominios básicos, los dominios coordinados por zinc, las hélices-giro-hélice y los factores beta-andamio.
Ver también
- Receptor nuclear, un tipo de factor de transcripción que se activa por moléculas específicas.
- Proteína G.
- Receptores en la Transcripción de Genes.
Véase también
 En inglés: Transcription factor Facts for Kids
En inglés: Transcription factor Facts for Kids