AlphaFold para niños
Datos para niños AlphaFold |
||
|---|---|---|
 |
||
| Información general | ||
| Tipo de programa | Modelo de inteligencia artificial Predicción de la estructura de las proteínas |
|
| Desarrollador | Google DeepMind | |
| Modelo de desarrollo | Código abierto | |
| Lanzamiento inicial | 2018 | |
| Licencia | Apache 2.0 License | |
| Información técnica | ||
| Programado en | Python | |
| Versiones | ||
| Última versión estable | 2.3.25 de abril de 2023 | |
| Enlaces | ||
|
Sitio web oficial
Repositorio de código
|
||
AlphaFold es un programa de inteligencia artificial (IA) creado por DeepMind, una empresa de Alphabet (la misma que Google). Su función principal es predecir cómo se doblan las proteínas, usando una técnica llamada aprendizaje profundo.
La primera versión de AlphaFold, llamada AlphaFold 1, ganó una competencia importante en diciembre de 2018. Esta competencia se llama CASP (Evaluación Crítica de Técnicas para la Predicción de la Estructura de Proteínas). AlphaFold 1 fue especialmente bueno prediciendo estructuras de proteínas que nunca antes se habían visto.
AlphaFold 2, que salió en 2020, también ganó la competencia CASP. Sus predicciones fueron mucho más precisas que las de cualquier otro programa. En la mayoría de los casos, AlphaFold 2 logró una puntuación muy alta, lo que significa que sus predicciones eran casi idénticas a las estructuras reales de las proteínas.
Los científicos han dicho que los resultados de AlphaFold 2 son "asombrosos" y "transformadores". Aunque no es perfecto, este avance es muy importante para la ciencia. En julio de 2021, la revista Nature publicó un artículo sobre AlphaFold 2, y también se lanzó su programa como software de código abierto y una base de datos con muchas estructuras de proteínas.
Contenido
¿Qué es el plegamiento de las proteínas?
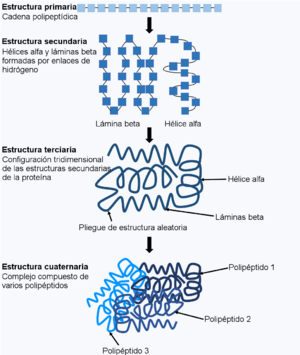
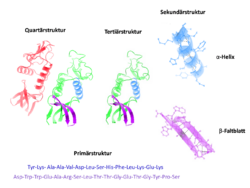
Las proteínas son como pequeñas máquinas en nuestro cuerpo que hacen muchas funciones importantes. Están hechas de largas cadenas de aminoácidos. Para funcionar correctamente, estas cadenas deben doblarse y enrollarse de una forma muy específica, creando una estructura tridimensional (3-D). Este proceso se llama plegamiento de proteínas.
El "problema del plegamiento de las proteínas" es un gran desafío para los científicos. Quieren entender cómo la secuencia de aminoácidos (el orden en que están los "ladrillos" de la proteína) decide la forma final en 3-D. Es como intentar saber la forma de un objeto solo conociendo la lista de sus piezas.
Normalmente, para saber la forma de una proteína, los científicos usan métodos especiales en laboratorios. Estos métodos son caros y tardan mucho tiempo. Se han descubierto las estructuras de unas 170.000 proteínas, pero se calcula que existen más de doscientos millones. Imagina lo útil que sería poder predecir la forma de una proteína solo conociendo su secuencia de aminoácidos.
Antes de AlphaFold, los programas de computadora no eran muy precisos. Solo podían predecir bien las estructuras de proteínas pequeñas y sencillas. AlphaFold, con su inteligencia artificial y aprendizaje profundo, cambió esto.
¿Cómo funciona AlphaFold?
DeepMind entrenó a AlphaFold usando información de más de 170.000 proteínas que ya se conocían. El programa usa una técnica de aprendizaje profundo que le permite identificar partes de un problema grande y luego unirlas para encontrar la solución completa. Para entrenar el programa, se usaron muchas computadoras potentes durante varias semanas. Una vez entrenado, AlphaFold puede predecir la estructura de una proteína en unos pocos días.
AlphaFold 1 (2018)
AlphaFold 1 se basó en la idea de que si dos partes de una proteína cambian juntas en diferentes especies, es probable que estén cerca una de la otra en la estructura final. El programa usó esta idea para calcular las distancias probables entre los aminoácidos y así predecir la forma de la proteína.
AlphaFold 2 (2020)
El equipo de DeepMind mejoró AlphaFold 1. Se dieron cuenta de que la primera versión a veces exageraba las interacciones entre aminoácidos que estaban cerca en la secuencia. AlphaFold 2 es un sistema más avanzado que aprende de forma integrada. Es como si el programa fuera armando un rompecabezas: primero une piezas pequeñas (grupos de aminoácidos) y luego busca cómo unir esos grupos para formar el todo.
Este proceso se repite varias veces, mejorando la predicción en cada paso. Al principio, la predicción puede tener algunos errores, pero con cada repetición, la forma se vuelve más precisa y correcta.
Competencias de predicción
CASP13
En diciembre de 2018, AlphaFold 1 fue el mejor en la competencia CASP13. Fue especialmente bueno prediciendo estructuras difíciles, donde no había modelos previos similares. AlphaFold predijo correctamente 25 de 43 proteínas en esta categoría. Su puntuación promedio fue de 58.9, superando a otros equipos.
Más tarde, en enero de 2020, parte del código de AlphaFold 1 se hizo público, pero solo para usarlo con los datos de esa competencia específica.
CASP14
En noviembre de 2020, AlphaFold 2, la nueva versión de DeepMind, ganó CASP14. Obtuvo los mejores resultados en 88 de 97 predicciones.
Su puntuación promedio fue de 92.4 sobre 100, lo que significa que sus predicciones eran casi tan precisas como las que se obtienen con métodos experimentales en laboratorio. Esto fue un gran salto, ya que dos años antes, AlphaFold 1 solo había logrado este nivel de precisión en dos predicciones.
Los modelos de AlphaFold 2 incluso ayudaron a descubrir la estructura de cuatro proteínas que los científicos no habían podido descifrar antes con otros métodos. Una de ellas era la proteína Af1503, que se había estudiado durante diez años.
Reacciones de los científicos
AlphaFold 2 es considerado un logro muy importante en la biología computacional. Venki Ramakrishnan, un científico que ganó el Premio Nobel, dijo que los resultados son "un avance asombroso" y que "cambiará fundamentalmente la investigación biológica".
Muchos medios de comunicación, tanto científicos como generales, hablaron de AlphaFold 2. Se comentó que la capacidad de predecir estructuras de proteínas con tanta precisión ayudará mucho en la investigación de enfermedades y en el desarrollo de nuevos medicamentos.
Transparencia y acceso
Una de las críticas a AlphaFold es que no se han compartido todos los detalles de cómo funciona. Aunque DeepMind hizo una presentación, no fue muy detallada. Algunos científicos han expresado que, aunque es un gran logro, sería más beneficioso para la ciencia si el programa fuera completamente abierto y accesible para todos.
Sin embargo, el hecho de que DeepMind haya logrado esto puede inspirar a otros equipos a desarrollar sistemas de IA similares. Es importante recordar que el éxito de AlphaFold 2 fue posible gracias a décadas de trabajo de muchos científicos que compartieron sus descubrimientos en bases de datos públicas.
También se ha señalado que AlphaFold necesita una infraestructura computacional muy grande, lo que podría limitar a otros grupos de investigación.
Predicciones de complejos y otras moléculas
AlphaFold 2 fue diseñado principalmente para predecir la estructura de proteínas individuales. No está tan claro qué tan bien funciona para proteínas que se unen a otras proteínas o a otras moléculas. Esto es importante porque en las células, las proteínas a menudo trabajan en equipo.
Además, AlphaFold solo se enfoca en las proteínas y no considera otras moléculas que pueden estar unidas a ellas, como metales o azúcares. Estas moléculas pueden ser muy importantes para la forma y función de la proteína. Los científicos sugieren que la base de datos de AlphaFold debería incluir esta información en el futuro.
Descubrimiento de medicamentos
Para que un modelo de proteína sea útil en el desarrollo de nuevos medicamentos, debe ser extremadamente preciso. Las predicciones de AlphaFold 2 son muy buenas, pero aún no son lo suficientemente perfectas para diseñar medicamentos directamente.
Los expertos también señalan que el mayor desafío en el desarrollo de medicamentos no es solo conocer la estructura de una proteína, sino entender si esa proteína es el objetivo correcto para tratar una enfermedad sin causar efectos secundarios.
Base de datos de AlphaFold
El 22 de julio de 2021, se lanzó la base de datos de estructuras de proteínas generadas por AlphaFold (AlphaFold DB). Esta base de datos contiene predicciones de estructuras de casi todas las proteínas humanas y de veinte organismos modelo, sumando más de 365.000 proteínas.
En julio de 2022, la base de datos se actualizó con estructuras de aproximadamente 200 millones de proteínas de 1 millón de especies, cubriendo casi todas las proteínas conocidas en la Tierra.
Limitaciones de la base de datos
La base de datos de AlphaFold tiene algunas limitaciones:
- Solo predice proteínas individuales, no cómo se unen para formar complejos más grandes.
- Puede tener baja confianza para proteínas que no tienen una forma fija.
- No está diseñada para analizar cambios pequeños en las proteínas (mutaciones).
- Solo genera una forma de la proteína, aunque algunas proteínas pueden tener varias formas.
- No incluye otras moléculas que pueden estar unidas a la proteína, como metales o azúcares.
Aplicaciones de AlphaFold
AlphaFold se ha usado para predecir varias estructuras de proteínas del SARS-CoV-2, el virus que causa la COVID-19. Esto es muy útil para entender mejor cómo funciona el virus. Un equipo de científicos confirmó que la estructura de una proteína del virus, predicha por AlphaFold 2, era muy similar a la que se había determinado experimentalmente. Esta proteína ayuda al virus a salir de las células y se cree que participa en la respuesta del cuerpo a la infección.
Véase también
 En inglés: AlphaFold Facts for Kids
En inglés: AlphaFold Facts for Kids
- Folding@home
- Foldit
- Rosetta@home
- AlphaZero
- AlphaGo
- Lista de proyectos de inteligencia artificial