Predicción de la estructura de las proteínas para niños
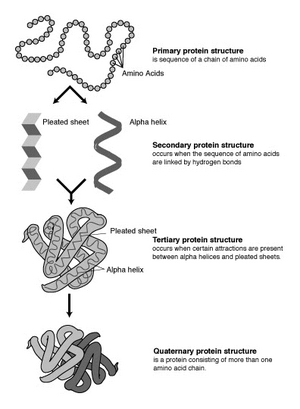
La predicción de la estructura de las proteínas es como intentar adivinar la forma tridimensional de una proteína basándose solo en la lista de sus "bloques de construcción", llamados aminoácidos. Imagina que tienes una cadena de cuentas de diferentes colores (los aminoácidos) y quieres saber qué forma compleja tomará esa cadena cuando se pliegue.
Este campo es muy importante en la bioinformática y la química teórica. Ayuda mucho en la medicina, por ejemplo, para diseñar fármacos que encajen perfectamente en una proteína específica. También es útil en la biotecnología para crear nuevas enzimas que hagan trabajos especiales.
Existen dos maneras principales de intentar predecir estas formas:
- Predicción desde cero (de novo): Se intenta construir la forma sin usar modelos previos, como si la proteína se plegara por primera vez.
- Predicción por comparación: Se busca en una biblioteca de formas de proteínas ya conocidas para ver si alguna se parece a la que queremos predecir.
Cada dos años, se realiza un evento llamado CASP (Evaluación Crítica de Técnicas para la Predicción de la Estructura de las Proteínas) para ver qué tan bien funcionan los métodos actuales. En julio de 2021, DeepMind y el EMBL lograron predecir más de 350,000 formas tridimensionales de proteínas, lo cual fue un gran avance.
Contenido
¿Qué es la Predicción de la Estructura Secundaria de las Proteínas?
La predicción de la estructura secundaria busca adivinar las formas locales que toma una cadena de proteína o ARN. Estas formas son como patrones básicos que se repiten. Para las proteínas, se trata de saber si una parte de la cadena formará una hélice alfa (como un muelle), una hebra beta (como una hoja plegada) o un bucle (una curva).
El éxito de estas predicciones se mide comparándolas con lo que se observa en la realidad, usando técnicas como la Cristalografía de rayos X. Para el ARN, se mira cómo se forman los puentes de hidrógeno. También se han creado programas para encontrar patrones especiales, como las hélices que atraviesan las membranas de las células.
Los mejores métodos actuales para predecir la estructura secundaria de las proteínas aciertan alrededor del 80% de las veces. Esto es muy útil para otras tareas, como clasificar los tipos de estructuras o mejorar la forma en que se comparan las secuencias de proteínas. La precisión de estos métodos se evalúa en comparaciones semanales como LiveBench y EVA.
¿Cómo Empezó la Predicción de la Estructura Secundaria?
Los primeros métodos para predecir la estructura secundaria surgieron en los años 60 y 70. Al principio, se enfocaban en encontrar las hélices alfa. Luego, en los años 70, aparecieron métodos más precisos que también incluían las hojas beta. Estos métodos se basaban en estadísticas, usando información de estructuras de proteínas ya conocidas.
Cuando se aplicaban a una sola secuencia, estos métodos tenían una precisión de alrededor del 60-65%. Sin embargo, a menudo no predecían bien las hojas beta. Se descubrió que si se analizaban varias secuencias de proteínas similares al mismo tiempo, la precisión mejoraba mucho.
Hoy en día, combinando estos métodos con grandes bases de datos de estructuras de proteínas y técnicas de aprendizaje automático (como las redes neuronales artificiales), se puede alcanzar hasta un 80% de precisión en las proteínas globulares. El límite máximo de precisión es de aproximadamente el 90%, porque hay pequeñas variaciones en las estructuras reales que son difíciles de predecir. Además, la predicción de la estructura secundaria no puede tener en cuenta la forma tridimensional completa de la proteína, lo que a veces limita su exactitud.
Métodos Clásicos de Predicción
Método de Chou-Fasman
El método de Chou-Fasman fue uno de los primeros algoritmos para predecir la estructura secundaria. Se basa en la probabilidad de que cada aminoácido aparezca en un tipo específico de estructura secundaria. Aunque ha sido actualizado, los resultados de este método son menos precisos que los de los métodos modernos, con una exactitud de aproximadamente el 50-60%.
Método GOR
El método GOR, nombrado por los científicos Garnier, Osguthorpe y Robson, se desarrolló poco después del Chou-Fasman. Utiliza la teoría de la información y probabilidades para predecir no solo la probabilidad de que un aminoácido tenga una estructura secundaria particular, sino también cómo influyen los aminoácidos vecinos. Este método es más preciso, alcanzando aproximadamente el 65% de exactitud, y es mejor prediciendo hélices alfa que hojas beta.
Aprendizaje Automático en la Predicción
Los métodos que usan redes neuronales artificiales aprenden de ejemplos de estructuras de proteínas ya conocidas para identificar patrones de secuencias asociados con ciertas estructuras secundarias. Estos métodos superan el 70% de precisión.
Las máquinas de soporte vectorial (MSV) son muy útiles para predecir la ubicación de los bucles, que son difíciles de identificar con otros métodos. Una ventaja de las MSV es que necesitan menos datos de entrenamiento.
Algunas técnicas de aprendizaje automático también intentan predecir detalles más finos de las proteínas, como los ángulos de su esqueleto.
Otras Mejoras en la Predicción Secundaria
La forma de una estructura secundaria no solo depende de la secuencia de aminoácidos. También influyen otros factores, como el ambiente local de la proteína, si está expuesta a un disolvente, el tipo de proteína y el organismo del que proviene. Por eso, añadir información sobre estos factores puede mejorar la predicción.
Los métodos de covarianza de secuencia analizan cómo cambian los aminoácidos en secuencias de ARN similares a lo largo de la evolución. Si dos partes de la secuencia que están lejos se mantienen unidas, esto puede indicar que hay un puente de hidrógeno importante para la estructura.
¿Qué es la Predicción de la Estructura Terciaria de las Proteínas?
La predicción de la estructura tridimensional completa de una proteína es más importante que nunca. Proyectos como el Proyecto Genoma Humano generan muchísimas secuencias de proteínas. Sin embargo, determinar la forma real de estas proteínas en el laboratorio (usando técnicas como la cristalografía de rayos X o la espectroscopia RMN) es muy lento y costoso. Por eso, la predicción por computadora es clave.
Predecir la estructura tridimensional de una proteína sigue siendo un desafío enorme. Los dos problemas principales son calcular la energía de la proteína y encontrar la forma que tiene la energía más baja. Un método de predicción debe explorar una cantidad inmensa de formas posibles.
Estos problemas se pueden simplificar con métodos como el modelado por homología y el enhebrado de proteínas, que asumen que la proteína que se estudia tiene una forma similar a otra proteína ya conocida. Los métodos de predicción desde cero (de novo o ab initio) no usan modelos previos y deben resolver estos problemas por sí mismos.
Modelado Ab Initio (Desde Cero)
Los métodos de modelado ab initio intentan construir modelos de proteínas desde cero, basándose en principios físicos. Algunos intentan imitar cómo se pliega una proteína en la naturaleza, mientras que otros usan métodos aleatorios para buscar posibles soluciones. Estos procesos requieren mucha capacidad de cálculo, por lo que solo se han usado para proteínas pequeñas. Predecir la estructura de una proteína grande desde cero necesita algoritmos mejores y computadoras muy potentes, como las supercomputadoras o la computación distribuida (proyectos como Folding@home o Rosetta@home).
Como paso intermedio para predecir estas estructuras, se usan los mapas de contacto, que muestran la distancia entre cada par de aminoácidos en una estructura tridimensional.
Modelado Comparativo
El modelado comparativo de proteínas usa estructuras ya conocidas como punto de partida. Es efectivo porque, aunque hay millones de proteínas diferentes, parece que solo existen unos 2000 tipos de formas básicas de plegamiento en la naturaleza.
Estos métodos se dividen en dos grupos:
- Modelado por homología: Se basa en la idea de que dos proteínas similares (homólogas) tendrán formas muy parecidas. Como la forma de una proteína se conserva más a lo largo de la evolución que su secuencia de aminoácidos, se puede modelar una proteína con buena precisión usando una plantilla de otra proteína lejanamente relacionada, siempre que se pueda ver la similitud en sus secuencias.
- Enhebrado de proteínas: Se compara la secuencia de aminoácidos de una proteína desconocida con una base de datos de estructuras ya resueltas. Se usa una puntuación para ver qué tan bien encaja la secuencia en cada estructura, obteniendo así posibles modelos tridimensionales. Este método también se conoce como "reconocimiento del plegado 3D-1D".
Predicción de la Geometría de las Cadenas Laterales
Predecir la forma exacta en que se organizan las cadenas laterales (las partes de los aminoácidos que sobresalen) es otro desafío. Se usan métodos que intentan encontrar la configuración de cadenas laterales con la energía más baja.
Para esto, se utilizan "bibliotecas de rotámeros", que son colecciones de formas favorables para las cadenas laterales de cada tipo de aminoácido. Estas bibliotecas se basan en el análisis de estructuras de proteínas conocidas.
Estos métodos son más útiles para analizar proteínas con un "núcleo" hidrófobo, donde las cadenas laterales están muy juntas. Tienen más dificultad con las cadenas laterales en la superficie, que son más flexibles.
Predicción de Clases Estructurales
También se han desarrollado métodos estadísticos para predecir a qué "clase" estructural pertenece una proteína, basándose en su composición de aminoácidos y otras características.
¿Qué es la Predicción de la Estructura Cuaternaria?
Cuando dos o más proteínas se unen para formar un complejo, y sus estructuras individuales son conocidas o pueden predecirse, se usan métodos de predicción de acoplamiento proteína-proteína para calcular la forma de ese complejo. Entender cómo se unen ayuda a diseñar nuevas moléculas que puedan interactuar con ellas.
Programas y Herramientas para la Predicción de Estructuras
Existen muchos programas y herramientas que ayudan en la predicción de estructuras de proteínas:
- MODELLER: Una herramienta popular para crear modelos por homología.
- SwissModel: Un servicio web automático para modelado por homología.
- I-TASSER: Fue el mejor programa para predicción de estructuras en los eventos CASP7 y CASP8.
- HHpred/HHsearch, bioinfo.pl, Robetta y Phyre: Herramientas comunes para el enhebrado de proteínas.
- RAPTOR (software): Un programa para enhebrado de proteínas.
- Abalone: Un programa para simular el plegamiento de proteínas.
- TIP: Una base de datos de modelos y similitudes entre secuencias y estructuras.
- Proyectos de computación distribuida: Como Folding@home, Rosetta@home, Human Proteome Folding Project, Predictor@home y TANPAKU, que usan la capacidad de cálculo de miles de computadoras de voluntarios para resolver problemas de plegamiento.
- Foldit: Un juego que permite a las personas resolver rompecabezas de plegamiento de proteínas, con el objetivo de mejorar el software de predicción.
- AlphaFold: Un programa de inteligencia artificial (IA) desarrollado por DeepMind (de Google) que usa el aprendizaje profundo para predecir estructuras de proteínas. AlphaFold 1 (2018) y AlphaFold 2 (2020) lograron los mejores resultados en CASP13 y CASP14, respectivamente.
Servidores para la Predicción Automática de Estructuras
El CASP (Evaluación Crítica de Técnicas para la Predicción de la Estructura de Proteínas) es un experimento que se realiza cada dos años desde 1994. Permite a investigadores y usuarios evaluar la calidad de los métodos y programas automáticos para predecir estructuras de proteínas.
Véase también
 En inglés: Protein structure prediction Facts for Kids
En inglés: Protein structure prediction Facts for Kids
- SIMAP