SiRNA para niños
siRNA son las siglas en inglés de small interfering RNA, que en español significa ARN pequeño de interferencia (ARNpi) o ARN de silenciamiento. Es un tipo de ARN especial que tiene dos cadenas, como una escalera de caracol.
Estos ARNpi miden entre 20 y 25 nucleótidos (los "ladrillos" que forman el ARN). Son muy específicos y se unen a un tipo de ARN llamado ARN mensajero (ARNm). Al unirse, el ARNpi interfiere con el mensaje del ARNm, lo que hace que un gen específico se "apague" o reduzca su actividad. Este proceso se llama interferencia de ARN (RNAi).
Los ARNpi también ayudan en otras funciones importantes, como defender a las células de los virus o ayudar a organizar el ADN dentro de la célula. El descubrimiento de cómo funcionan estas moléculas fue tan importante que Craig C. Mello y Andrew Fire ganaron el Premio Nobel de Fisiología o Medicina en 2006 por ello.

Contenido
¿Qué son los ARNpi y cómo funcionan?
Los ARNpi son moléculas de ARN de doble cadena, de unos 20 a 25 nucleótidos de largo. Se forman a partir de moléculas de ARN de doble cadena más grandes (llamadas dsRNA). Estos dsRNA pueden venir de dentro de la célula (por ejemplo, de partes repetidas del ADN) o de fuera (como de virus o genes introducidos artificialmente).
La enzima principal que corta los dsRNA en ARNpi es Dicer. Esta enzima se encuentra en el citoplasma de la célula y corta los dsRNA en pedacitos de ARNpi.
Cómo los ARNpi "apagan" los genes
Los ARNpi funcionan "apagando" la actividad de los genes. Lo hacen cortando el ARN mensajero (ARNm) que lleva las instrucciones para fabricar proteínas. Esto ocurre cuando una de las cadenas del ARNpi se une a un grupo de proteínas llamado complejo RISC (RNA-induced silencing complex).
Una vez que el ARNpi y el complejo RISC se unen, buscan el ARNm que coincide con la secuencia del ARNpi. Cuando lo encuentran, el complejo RISC corta el ARNm en dos pedazos. Después, la célula destruye esos pedazos de ARNm. Al no haber ARNm, el gen no puede dar sus instrucciones y su actividad se reduce.
Además, los ARNpi también pueden ayudar a modificar el ADN, haciendo que ciertas partes de los cromosomas se vuelvan más compactas y menos activas.
El descubrimiento de la interferencia de ARN
Observaciones en plantas
El mecanismo de interferencia de ARN (RNAi) se vio por primera vez en plantas. Los científicos notaron que, al intentar cambiar el color de las flores de petunias, a veces las flores salían con manchas blancas. Esto significaba que los genes que daban color a la flor se estaban "apagando". Este efecto se llamó co-supresión.
Se dieron cuenta de que esto podía pasar por varias razones, como la presencia de genes extraños o infecciones por virus. Se pensó que era un sistema de defensa de las plantas contra los virus, que destruía el ARN de los virus. Sin embargo, no se sabía exactamente cómo funcionaba.
Experimentos en gusanos

En la década de 1990, otros científicos trabajaban con un pequeño gusano llamado C. elegans. Intentaban "apagar" genes inyectando un tipo de ARN llamado ARN-antisentido. Pensaban que este ARN se uniría al ARNm y evitaría que se fabricaran proteínas.
Sin embargo, dos investigadores, Guo y Kemphues, descubrieron algo sorprendente: inyectar ARN-sentido (el "gemelo" del ARN-antisentido) también "apagaba" los genes. No entendían por qué.
En 1998, Andrew Fire y Craig C. Mello publicaron un estudio clave. Demostraron que si inyectaban solo una cadena de ARN (sentido o antisentido) purificada, no pasaba nada. Pero si inyectaban ARN de doble cadena (dsRNA), el gen se "apagaba" de forma muy específica. Descubrieron que los experimentos anteriores de Guo y Kemphues habían usado ARN no purificado, que contenía pequeñas cantidades de ARN de doble cadena, y eran estas las que causaban el efecto.
Fire y Mello también demostraron que la interferencia ocurría a nivel del ARNm: cuando inyectaban dsRNA, el ARNm del gen objetivo desaparecía. Esto confirmaba que el proceso "apagaba" el gen después de que se copiara el mensaje (post-transcripcional).
Además, vieron que el efecto de "apagado" no solo ocurría en el lugar de la inyección, sino en la mayoría de los tejidos del gusano. Esto sugería que la señal podía viajar de una célula a otra.
La prueba de los ARNpi
Una vez que se supo que el RNAi actuaba sobre el ARNm, la pregunta era: ¿qué causaba la degradación del ARNm?
En 1999, Hamilton y Baulcombe realizaron un experimento con plantas de tomate. Descubrieron que en las plantas donde un gen estaba "apagado", aparecían pequeñas moléculas de ARN de unos 25 nucleótidos. Estas moléculas eran tanto de la cadena sentido como de la antisentido. Esto demostró la existencia de estos pequeños ARN, que no eran solo productos de la degradación del ARNm.

Para entender mejor el RNAi, se usaron sistemas en el laboratorio con embriones de la mosca Drosophila. En 2000, Zamore, Tuschl y sus colegas demostraron que el ARNm se degradaba si se le añadía dsRNA. También vieron que los dsRNA se cortaban en pequeños fragmentos de 21 a 23 nucleótidos, tanto la cadena sentido como la antisentido.
Así, se concluyó que los fragmentos de dsRNA (los ARNpi) eran los que reconocían el ARNm y causaban su degradación.
Los componentes del RNAi
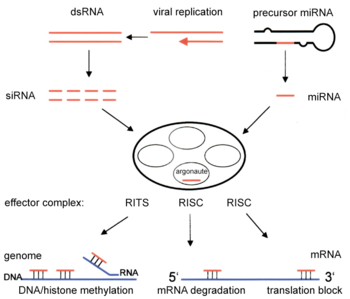
Estudios posteriores revelaron más detalles. Cuando un organismo recibe ARN de doble cadena (por ejemplo, de un virus), una enzima llamada Dicer lo reconoce y lo corta en fragmentos de unos 22 nucleótidos. Estos pequeños dsRNA (los ARNpi) se unen a un complejo llamado RISC (RNA-induced silencing complex).
El complejo RISC se activa y usa una de las cadenas del ARNpi (la cadena antisentido) como guía para encontrar el ARNm que coincide. Luego, el RISC corta y destruye el ARNm, "apagando" el gen.
Identificación de Dicer
Los científicos buscaron la enzima que cortaba los dsRNA. En 2001, Bernstein y sus colegas demostraron que solo la enzima Dicer era capaz de cortar los dsRNA largos en los pequeños fragmentos de unos 22 nucleótidos.
El complejo RISC
Los ARNpi creados por Dicer se unen al complejo RISC, que contiene varias proteínas. La parte más importante del RISC es una proteína llamada Argonauta. Esta proteína es la que corta el ARNm.
De las dos cadenas del ARNpi, solo una (la cadena antisentido) se une a la proteína Argonauta y guía el proceso de degradación del ARNm. La otra cadena (la sentido) se desecha.
Mecanismo de interferencia de ARN (RNAi) mediado por ARNpi
El proceso de RNAi comienza cuando una célula detecta un ARN de doble cadena largo. La enzima Dicer lo corta en ARNpi. Luego, un complejo llamado RISC elige una de las cadenas del ARNpi (la guía) y la usa para encontrar el ARNm que coincide.
En organismos como la mosca Drosophila y los mamíferos, la cadena guía del ARNpi se une al complejo RISC activado. Este complejo puede degradar muchas moléculas del ARNm objetivo, "apagando" el gen. Generalmente, el "apagado" no es total, sino que reduce la actividad del gen.
En plantas y gusanos, el proceso es un poco diferente. Después de que Dicer corta el dsRNA, la cadena guía del ARNpi puede unirse a una enzima especial que crea nuevas moléculas de dsRNA. Dicer vuelve a cortar estas nuevas moléculas, creando más ARNpi. Esto amplifica la señal y hace que el efecto de "apagado" se extienda por todo el organismo, ayudando a defenderse de infecciones.
Amplificación de la señal
Un descubrimiento interesante fue que en gusanos, el efecto de "apagado" podía extenderse a otros genes relacionados. Esto se debe a la enzima ARN polimerasa-dependiente de ARN (RdRP), que usa los ARNpi como "cebador" para crear más ARN de doble cadena. Esto amplifica la señal y puede afectar a genes cercanos.
Relación entre la cantidad de ARN y el efecto
Se observó que una pequeña cantidad de ARN de doble cadena era mucho más efectiva para "apagar" genes que una gran cantidad de ARN de una sola cadena. Esto sugería que el ARN de doble cadena actuaba como un catalizador, es decir, que unas pocas moléculas podían iniciar un proceso que se amplificaba, "apagando" muchos genes.
Resumen del RNAi mediado por ARNpi
En resumen, un ARN de doble cadena largo es cortado por Dicer en ARNpi. La cadena antisentido del ARNpi se une al complejo RISC, que se activa. Este complejo encuentra el ARNm que coincide con el ARNpi y lo corta, haciendo que el gen se "apague".
Este "apagado" de genes es muy específico y solo afecta al ARNm que coincide con la secuencia del ARNpi. El resultado es una reducción significativa o completa de la actividad del gen.
En plantas, este proceso se llama "silenciamiento génico post-transcripcional" (PTGS). Estos mecanismos son importantes para la defensa contra virus y otros elementos genéticos.
El pequeño tamaño de los ARNpi permite que se muevan entre células, lo que explica los efectos que se observan en todo el organismo.
Uso en el laboratorio
Herramientas para "apagar" genes
Los científicos usan diferentes herramientas para aplicar el RNAi en el laboratorio, dependiendo del organismo:
- En C. elegans (gusanos), se usan dsRNA largos que se pueden inyectar o incluso dar a los gusanos en su comida.
- En Drosophila melanogaster (moscas), también se usan dsRNA largos, inyectándolos en los embriones o haciendo que las células los produzcan.
- En plantas, se pueden introducir genes que produzcan dsRNA.
- En mamíferos (como ratones o células humanas en cultivo), se pueden inyectar ARNpi sintéticos o usar virus modificados para que las células produzcan los ARNpi.
RNAi en mamíferos
Aunque los dsRNA largos pueden "apagar" genes en las primeras etapas de desarrollo de mamíferos, en las células adultas pueden activar una respuesta de defensa que puede dañar la célula.
Sin embargo, en 2001, Thomas Tuschl y sus colegas descubrieron que los ARNpi sintéticos de menos de 30 nucleótidos podían "apagar" genes en células de mamíferos sin activar esa respuesta de defensa. Este descubrimiento fue muy importante para usar el RNAi en la investigación médica y para desarrollar nuevos tratamientos.
ARNpi y ARNsh
En mamíferos, se pueden usar ARNpi sintéticos o moléculas llamadas ARNsh (short hairpin RNA). Los ARNsh son como "horquillas" de ARN que la enzima Dicer corta para producir ARNpi funcionales.
Los ARNpi son muy específicos, pero a veces pueden "apagar" genes que no son el objetivo principal. Esto se llama "efecto fuera de objetivo" (off-target). Los científicos trabajan para reducir estos efectos modificando los ARNpi o usando concentraciones bajas.
Aplicaciones
La capacidad del RNAi para "apagar" genes de forma específica lo convierte en una herramienta muy valiosa en la investigación. Permite a los científicos entender la función de los genes. Por ejemplo, pueden "apagar" un gen y ver qué cambios ocurren en la célula o el organismo.
También se usa en la genómica funcional, que es el estudio de cómo funcionan todos los genes de un organismo. Esto ayuda a identificar qué genes son necesarios para procesos celulares específicos, como la división celular.
Además, el RNAi es una herramienta prometedora en la biotecnología y la medicina.
"Apagado" de genes (knockdown)
El RNAi se usa para reducir drásticamente la actividad de un gen. Esto se llama "knockdown" y es diferente de un "knockout", donde el gen se "apaga" por completo. Al estudiar los efectos de esta reducción, los científicos pueden entender qué hace la proteína que produce ese gen.
Genómica funcional
El RNAi es muy útil para estudiar genes en organismos modelo como C. elegans y Drosophila, donde es muy efectivo. También se están haciendo esfuerzos para usarlo en células de mamíferos.
En plantas, el RNAi es especialmente útil para estudiar genomas complejos, como el del trigo, que tiene muchas copias de sus genes.
Medicina
Una de las aplicaciones más emocionantes del RNAi es en la medicina. Aunque es difícil usar dsRNA largos en mamíferos, los ARNpi sintéticos o los ARNsh han tenido más éxito. Ya se están probando tratamientos para enfermedades como la degeneración macular (una enfermedad de los ojos) y algunas infecciones virales.
También se investiga su uso en tratamientos para enfermedades neurodegenerativas (como la enfermedad de Huntington) y para el cáncer, "apagando" genes que promueven el crecimiento de las células enfermas.
Un desafío importante es cómo llevar los ARNpi de forma segura a las células del cuerpo. Se están investigando diferentes métodos, como el uso de virus modificados o la encapsulación de los ARNpi en pequeñas esferas de grasa.
A pesar de su potencial, los científicos siguen investigando para asegurar que los tratamientos con RNAi sean seguros y evitar los "efectos fuera de objetivo" (off-target), donde se "apagan" genes no deseados.
Biotecnología
El RNAi también se ha usado en biotecnología, especialmente para mejorar plantas. Por ejemplo, se ha utilizado para reducir los niveles de sustancias no deseadas en plantas comestibles. Las semillas de algodón son ricas en proteínas, pero contienen una sustancia tóxica llamada gossypol. Usando RNAi, se han creado variedades de algodón cuyas semillas tienen menos gossypol, sin afectar la protección de la planta contra plagas.
También se han hecho esfuerzos para reducir otras sustancias no deseadas en plantas como la yuca. Aunque aún no hay muchos productos en el mercado que usen RNAi, se ha logrado reducir alérgenos en tomates y sustancias que podrían ser dañinas en plantas de tabaco. También se ha usado para hacer plantas más resistentes a virus.
Véase también
 En inglés: Small interfering RNA Facts for Kids
En inglés: Small interfering RNA Facts for Kids

